Wat is GLM in R?
Gegeneraliseerde lineaire modellen is een subset van lineaire regressiemodellen en ondersteunt niet-normale distributies effectief. Om dit te ondersteunen, wordt aanbevolen de functie glm () te gebruiken. GLM werkt goed met een variabele wanneer de variantie niet constant is en normaal wordt verdeeld. Een koppelingsfunctie is gedefinieerd om de responsvariabele te transformeren naar het juiste model. Een LM-model wordt gedaan met zowel de familie als de formule. GLM-model heeft drie belangrijke componenten genaamd random (waarschijnlijkheid), Systematic (lineaire voorspeller), linkcomponent (voor logit-functie). Het voordeel van het gebruik van glm is dat ze modelflexibiliteit hebben, geen constante variantie nodig heeft en dit model past bij de maximale waarschijnlijkheidsschatting en de bijbehorende verhoudingen. In dit onderwerp gaan we meer leren over GLM in R.
GLM-functie
Syntaxis: glm (formule, familie, gegevens, gewichten, subset, Start = null, model = WAAR, methode = ””…)
Hier zijn Family types (inclusief model types) binomial, Poisson, Gaussian, gamma, quasi. Elke distributie voert een ander gebruik uit en kan worden gebruikt in zowel classificatie als voorspelling. En wanneer het model Gaussisch is, moet de reactie een heel geheel getal zijn.
En wanneer het model binomiaal is, moet het antwoord klassen zijn met binaire waarden.
En wanneer het model Poisson is, moet de respons niet-negatief zijn met een numerieke waarde.
En wanneer het model gamma is, moet de respons een positieve numerieke waarde zijn.
glm.fit () - Om in een model te passen
Lrfit () - geeft een logische regressiefit aan.
update () - helpt bij het bijwerken van een model.
anova () - het is een optionele test.
Hoe GLM in R te maken?
Hier zullen we zien hoe we een eenvoudig gegeneraliseerd lineair model met binaire gegevens kunnen maken met behulp van de functie glm (). En door verder te gaan met de dataset van Trees.
Voorbeelden
// Een bibliotheek importerenlibrary(dplyr)
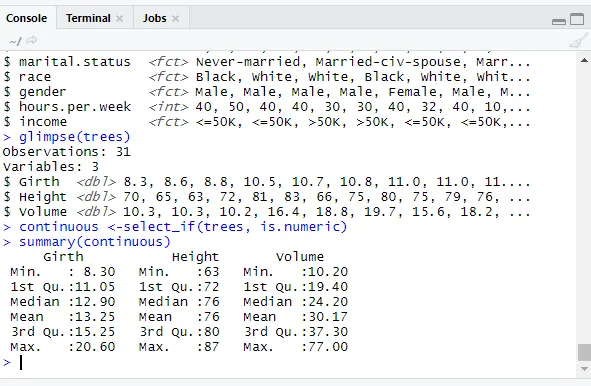
glimpse(trees)

Om categorische waarden te zien, worden factoren toegewezen.
levels(factor(trees$Girth))
// Doorlopende variabelen verifiëren
library(dplyr)
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Boomgegevensset opnemen in R-zoekopdracht Pathattach (bomen)
x<-glm(Volume~Height+Girth)
x
Output:
| Oproep: glm (formule = volume ~ hoogte + omtrek)
coëfficiënten: (Onderscheppen) Hoogteomtrek -57.9877 0.3393 4.7082 Vrijheidsgraden: 30 Totaal (dwz nul); 28 Rest Null Deviance: 8106 Restafwijking: 421.9 AIC: 176.9 |
summary(x)
| Oproep:
glm (formule = volume ~ hoogte + omtrek) Afwijkende residuen: Min 1Q Mediaan 3Q Max -6.4065 -2.6493 -0.2876 2.2003 8.4847 coëfficiënten: Schat Std. Fout t waarde Pr (> | t |) (Onderscheppen) -57.9877 8.6382 -6.713 2.75e-07 *** Hoogte 0.3393 0.1302 2.607 0.0145 * Omtrek 4.7082 0.2643 17.816 <2e-16 *** - Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0, 1 '' 1 (Dispersieparameter voor Gaussian familie genomen op 15.06862) Nul-afwijking: 8106.08 op 30 vrijheidsgraden Restafwijking: 421, 92 op 28 vrijheidsgraden AIC: 176, 91 Aantal Fisher Scoring iteraties: 2 |
De uitvoer van de samenvattingfunctie geeft de aanroepen, coëfficiënten en residuen weer. De bovenstaande respons geeft aan dat zowel de hoogte als de omtrekcoëfficiënt niet significant zijn, omdat de waarschijnlijkheid kleiner is dan 0, 5. En er is een variant van afwijking genaamd null en residual. Ten slotte is het scoren van vissers een algoritme dat maximale waarschijnlijkheidsproblemen oplost. Met binomiaal is de reactie een vector of matrix. cbind () wordt gebruikt om de kolomvectoren in een matrix te binden. En om de gedetailleerde informatie van de fit-samenvatting te krijgen, wordt gebruikt.
Om de Hood-test uit te voeren wordt de volgende code uitgevoerd.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
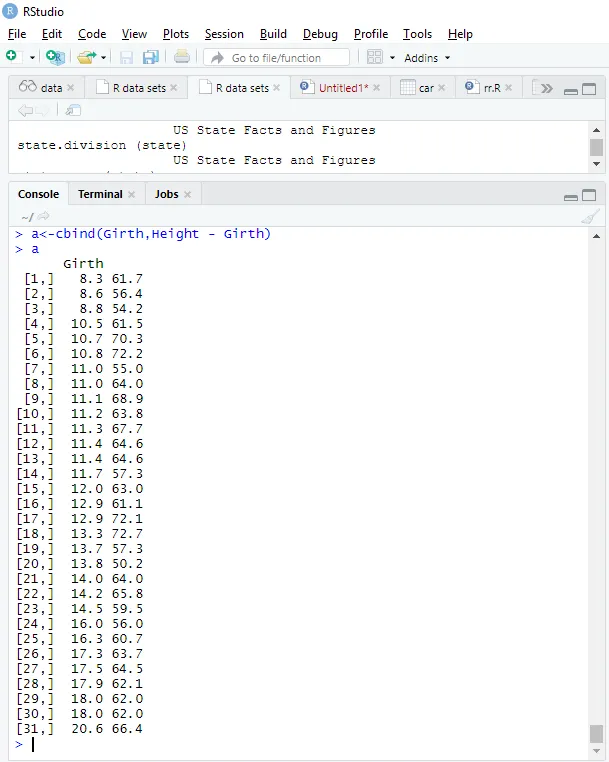
Model fit
a<-cbind(Height, Girth - Height)
> a
samenvatting (bomen)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
Om de juiste standaardafwijking te krijgen
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
Vervolgens verwijzen we naar de telresponsvariabele om een goede respons te modelleren. Om dit te berekenen, gebruiken we de dataset USAccDeath.
Laten we de volgende fragmenten in de R-console invoeren en kijken hoe de jaartelling en het jaarvierkant daarop worden uitgevoerd.
data("USAccDeaths")
force(USAccDeaths)
// Om het jaar 1973-1978 te analyseren.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Oproep:
glm (formule = aantal ~ jaar + jaarSqr, familie = "poisson", data = schijf) Afwijkende residuen: Min 1Q Mediaan 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 coëfficiënten: Schat Std. Fout z waarde Pr (> | z |) (Onderscheppen) 9.187e + 00 3.557e-03 2582.49 <2e-16 *** jaar -7.207e-03 2.354e-04 -30.62 <2e-16 *** jaarSqr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0, 1 '' 1 (Dispersieparameter voor Poisson-familie genomen als 1) Ongeldige afwijking: 7357.4 op 71 vrijheidsgraden Restafwijking: 6358.0 op 69 vrijheidsgraden AIC: 7149.8 Aantal Fisher Scoring iteraties: 4 |
Om te controleren of het model het beste past, kan het volgende commando worden gebruikt om te zoeken
de residuen voor de test. Uit het onderstaande resultaat is de waarde 0.
1 - pchisq(deviance(a1), df.residual(a1))
QuasiPoisson-familie gebruiken voor de grotere variantie in de gegeven gegevens
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Oproep:
glm (formule = aantal ~ jaar + jaarSqr, familie = "quasipoisson", data = disc) Afwijkende residuen: Min 1Q Mediaan 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 coëfficiënten: Schat Std. Fout t waarde Pr (> | t |) (Onderscheppen) 9.187e + 00 3.417e-02 268.822 <2e-16 *** jaar -7.207e-03 2.261e-03 -3.188 0.00216 ** jaarSqr 8.841e-05 3.095e-05 2.857 0.00565 ** - (Dispersieparameter voor quasipoisson-familie genomen als 92.28857) Ongeldige afwijking: 7357.4 op 71 vrijheidsgraden Restafwijking: 6358.0 op 69 vrijheidsgraden AIC: NA Aantal Fisher Scoring iteraties: 4 |
Het vergelijken van Poisson met binomiale AIC-waarde verschilt aanzienlijk. Ze kunnen worden geanalyseerd door precisie en recall-ratio. De volgende stap is om te controleren of de restvariantie evenredig is aan het gemiddelde. Vervolgens kunnen we plotten met behulp van de ROCR-bibliotheek om het model te verbeteren.
Conclusie
Daarom hebben we ons gericht op een speciaal model genaamd gegeneraliseerd lineair model dat helpt bij het focussen en schatten van de modelparameters. Het is vooral het potentieel voor een continue responsvariabele. En we hebben gezien hoe glm in een R-pakket past. Ze zijn de meest populaire benaderingen voor het meten van telgegevens en een robuust hulpmiddel voor classificatietechnieken die worden gebruikt door een gegevenswetenschapper. R-taal helpt natuurlijk bij het uitvoeren van gecompliceerde wiskundige functies
Aanbevolen artikelen
Dit is een gids voor GLM in R. Hier bespreken we de GLM-functie en hoe GLM in R te maken met voorbeelden en uitvoer van boomgegevenssets. U kunt ook het volgende artikel bekijken voor meer informatie -
- R Programmeertaal
- Big Data-architectuur
- Logistieke regressie in R
- Big Data Analytics-opdrachten
- Poisson-regressie in R | Poisson-regressie implementeren